Chapter 5 Visualización
Se procederá al análisis visual de los datos ya curados y normalizados. Se evaluarán las dos categorías indicativas del objeto “sexo” y “tratamiento”
#Calculo de factores de normalización
dge <- DGEList(
counts = assay(rse_gene_SRP162774, "counts"),
genes = rowData(rse_gene_SRP162774)
)
dge <- calcNormFactors(dge)La siguiente gráfica ilustra la distribución de expresión de ambas condiciones. Por lo que se puede ver que están encasilladas entre 0.1 a poco más de 0.21, ambas condiciones tienen expresión similar, sin embargo se puede percibir un grupo de outliers para la expresión de ibuprofeno
## Visualización de expresión por medio de boxplot
ggplot(as.data.frame(colData(rse_gene_SRP162774)), aes(y = assigned_gene_prop, x = sra_attribute.drug_treatment, fill=sra_attribute.drug_treatment)) +
geom_boxplot(alpha=0.3) +
theme_bw(base_size = 20) +
ylab("Proporción génica") +
xlab("Tratamiento") +
theme(legend.position="top", legend.title = element_text(size = 13), legend.text = element_text(size = 8)) +
labs(fill = "Relación de expresión génica con tipo de tratamiento |")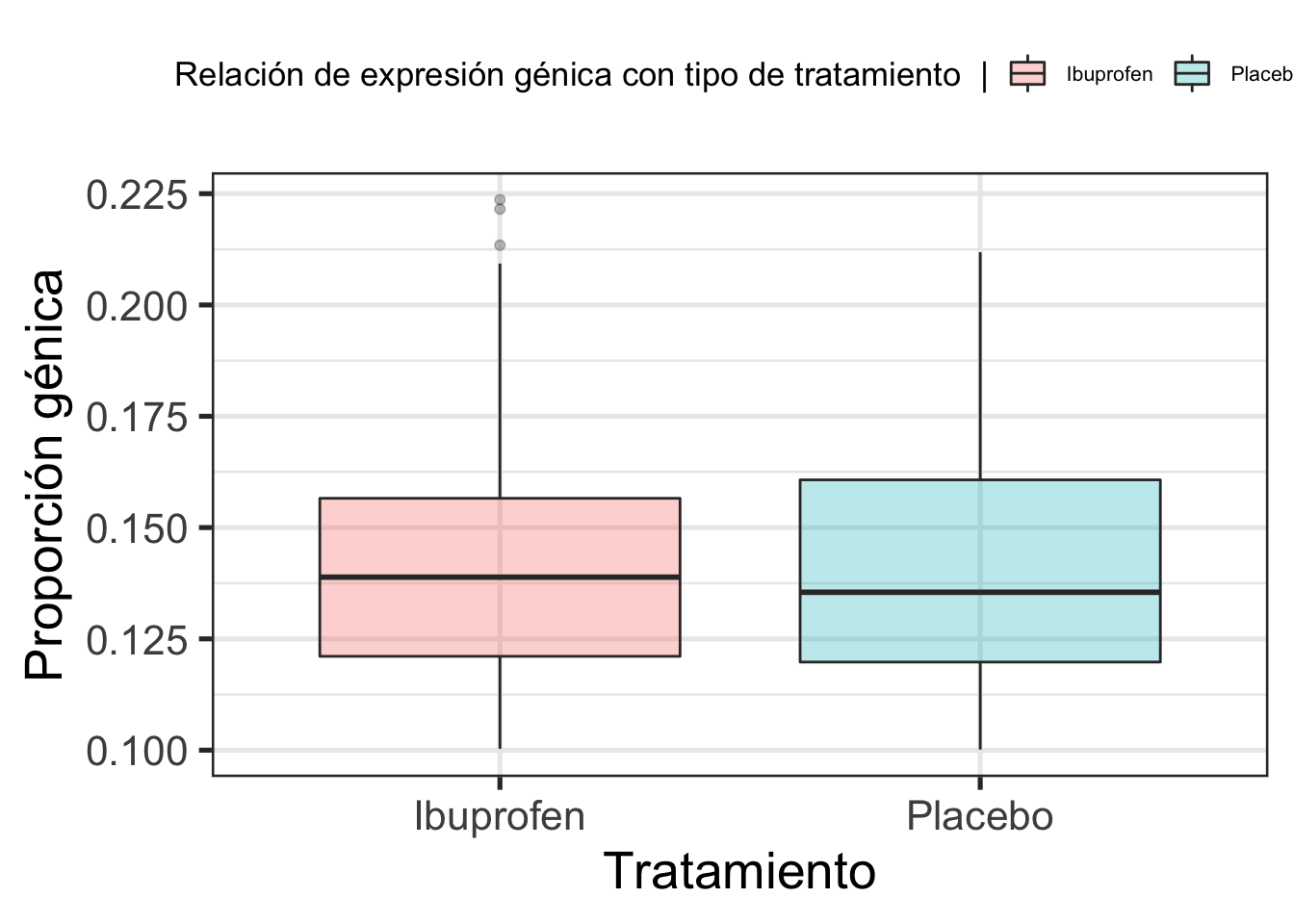
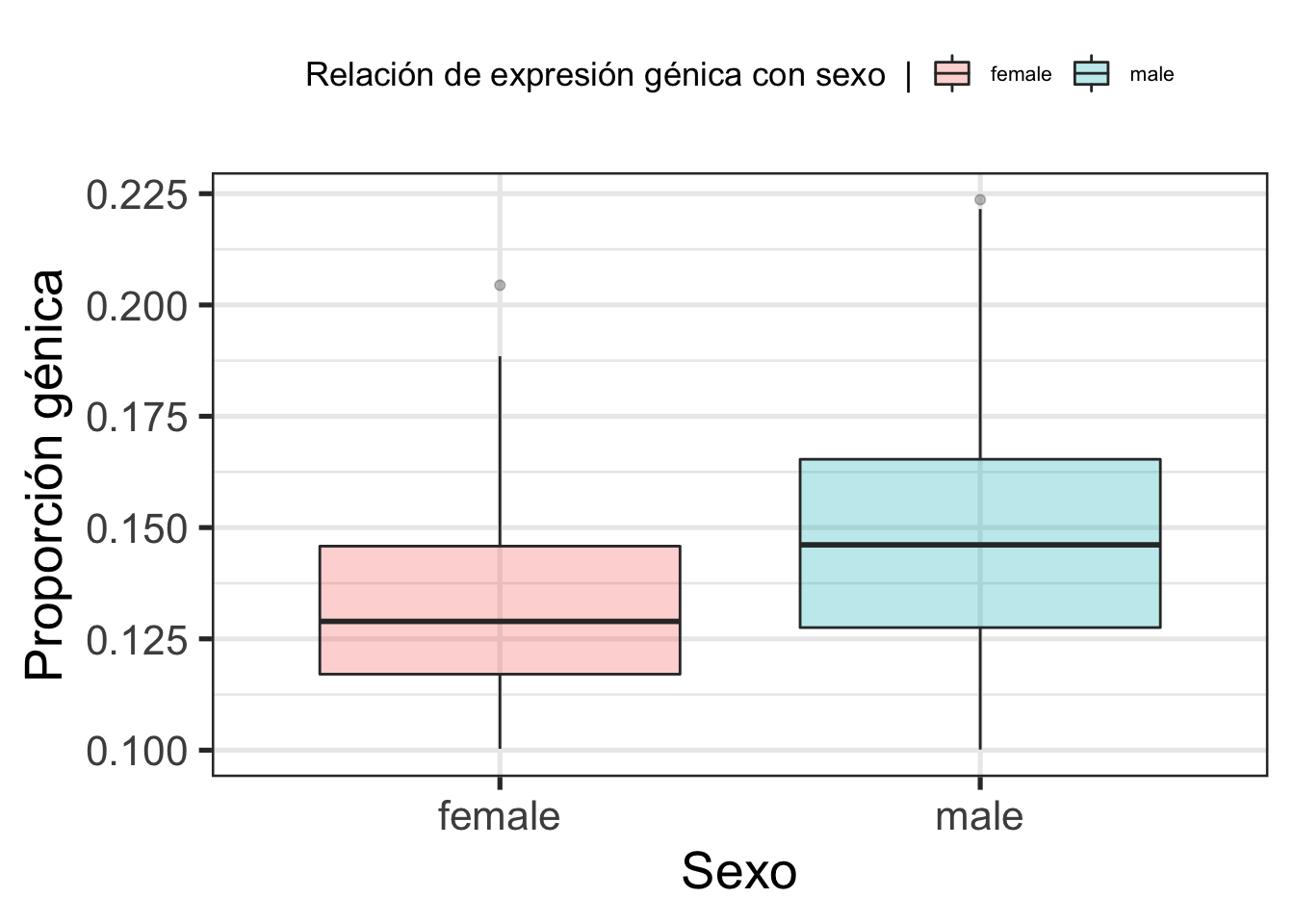
ggplot(as.data.frame(colData(rse_gene_SRP162774)), aes(y = assigned_gene_prop, x = sra_attribute.gender, fill=sra_attribute.gender)) +
geom_boxplot(alpha=0.3) +
theme_bw(base_size = 20) +
ylab("Proporción génica") +
xlab("Sexo") +
theme(legend.position="top", legend.title = element_text(size = 13), legend.text = element_text(size = 8)) +
labs(fill = "Relación de expresión génica con sexo |")